4 Linear Mixed Effects Models
4.1 Theorie in Kürze
- Lineare Regression mit fixen Effekten (übliche \(\beta\)’s) und zufälligen Effekten (gruppenspezifische Störgrössen); fixed + zufällig = mixed.
- Es werden die fixen Effekte und die Varianzen der zufälligen Effekte geschätzt; in diesem Prozess werden auch die modellierten zufälligen Effekte pro Person (bzw. Gruppe) gespeichert und können mit der Funktion
ranefabgerufen werden. - Lineare Regression verlangte immer unabhängige Beobachtungen; bei den Mixed Models dürfen Beobachtungen innerhalb einer Gruppe (z.B. mehrere Messungen pro Person) korreliert sein; als Faustregel gilt: Für jede Gruppierung braucht man einen entsprechenden zufälligen Effekt.
- Alternativ kann man pro Gruppierung eine Faktorvariable (“Blockfaktor”) in einer herkömmlichen Linearen Regression verwenden; beim Blockfaktor liegt der Fokus auf den Erwartungswerten der einzelnen Gruppen; beim Mixed Model liegt der Fokus der zu Grunde liegenden Population und der Streuung zwischen den einzelnen Gruppen.
- Einfache Mixed Effects Modelle sind: Random Intercept Modell (RI) und Random Intercept and Random Slope Modell (RIRS).
RI-Modell (Person \(i\), Beobachtung \(j\)); geschätzt werden \(\beta_0\), \(\beta_1\), \(\sigma\), \(\sigma_u\): \[\begin{array}{c} Y_{ij} = (\beta_0 + u_i) + \beta_1 x_j + \varepsilon_{ij}, \\ \varepsilon_{ij} \sim N(0, \sigma^2),\\ u_i \sim N(0, \sigma_u^2) \end{array}\]
RIRS-Modell: (Person \(i\), Beobachtung \(j\)); geschätzt werden \(\beta_0\), \(\beta_1\), \(\sigma\), \(\sigma_1, \sigma_2, \rho\): \[\begin{array}{c} Y_{ij} = (\beta_0 + u_{1,i}) + (\beta_1 + u_{2,i})x_j + \varepsilon_{ij}, \\ \varepsilon_{ij} \sim N(0, \sigma^2), \\ \quad u_{1,i} \sim N(0, \sigma_1^2), \\ \quad u_{2,i} \sim N(0, \sigma_2^2), \\ cor(u_1, u_2) = \rho \end{array}\]
Residuenanalyse wie bei Linearer Regression; zudem sollte man prüfen, ob die modellierten Zufallseffekte ungefähr normalverteilt sind
Wichtige R Funktionen im Paket
lme4bzw.lmerTest:lmer,summary,predict,qqnorm,confint,ranef.
4.2 Bsp: RIRS-Modell
In diesem Beispiel schätzen wir ein RIRS-Modell mit den Daten der
sleepstudy aus dem Paket lme4 (siehe ?sleepstudy für weitere
Infos dazu). Eine Umsetzung mit dem Paket nlme findet man weiter unten.
library(lme4)
library(lmerTest)
fm <- lmer(Reaction ~ Days + (Days | Subject), sleepstudy)
summary(fm)## Linear mixed model fit by REML. t-tests use Satterthwaite's method [
## lmerModLmerTest]
## Formula: Reaction ~ Days + (Days | Subject)
## Data: sleepstudy
##
## REML criterion at convergence: 1743.6
##
## Scaled residuals:
## Min 1Q Median 3Q Max
## -3.9536 -0.4634 0.0231 0.4634 5.1793
##
## Random effects:
## Groups Name Variance Std.Dev. Corr
## Subject (Intercept) 612.10 24.741
## Days 35.07 5.922 0.07
## Residual 654.94 25.592
## Number of obs: 180, groups: Subject, 18
##
## Fixed effects:
## Estimate Std. Error df t value Pr(>|t|)
## (Intercept) 251.405 6.825 17.000 36.838 < 2e-16 ***
## Days 10.467 1.546 17.000 6.771 3.26e-06 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Correlation of Fixed Effects:
## (Intr)
## Days -0.138## 2.5 % 97.5 %
## sd_(Intercept)|Subject 14.3814732 37.7159917
## cor_Days.(Intercept)|Subject -0.4815008 0.6849863
## sd_Days|Subject 3.8011643 8.7533851
## sigma 22.8982669 28.8579965
## (Intercept) 237.6806955 265.1295147
## Days 7.3586533 13.5759188Im Block Fixed effects sehen wir die geschätzten
Populations-Mittelwerte. Die Interpretation in diesem Block ist analog
zur Interpretation bei der linearen Regression. Die Zeile
(Intercept) zeigt die durchschnittliche Reaktionszeit einer
ausgeruhten Person (0 Tage Schlafentzug): \(251\) ms (95%-VI: \([237; 265]\) aus dem Output des Befehls ). Pro Tag
Schlafentzug nimmt die erwartete Reaktionszeit gemäss unserem Modell
um ca. \(10.5\) ms zu (95%-VI: \([7.4; 13.6]\)).
Im Block Random effects stehen die geschätzten Varianzen (und
evtl. Korrelation) der zufälligen Effekte und des Fehlers (in der
Zeile Residual). In diesem Fall haben wir eine Varianz für
den unterschiedlichen Achsenabschnitt von Person zu Person (Random
Intercept) und eine Varianz für die unterschiedliche Steigung von
Person zu Person (Random Slope). Diese Werte stehen in der Spalte
Variance. Daneben in der Spalte steht
einfach die Wurzel der Varianzen (Achtung: Hier steht
nicht der Standard Error wie wir es beim Output der linearen
Regression gewöhnt sind, sondern wirklich nur die Wurzel aus der
Spalte Variance). Die Standardabweichung (Spalte
Std.Dev.) der Random Intercepts ist also ca. \(24.7\) ms
(95%-VI: \([14.4; 37.7]\) ms). Eine typische Schwankung der mittleren
Reaktionszeit im ausgeruhten Zustand von Person zu Person ist also
ca. \(24.7\) ms. Die Standardabweichung der Schwankung bzgl. Steigung von
Person zu Person ist ca. \(5.9\) (95%-VI: \([3.8; 8.8]\)). D.h., die
Zunahme der Reationszeit pro Nacht mit Schlafentzug ist von Person zu
Person unterschiedlich. Eine typische Schwankung der Steigung von
Person zu Person ist ca. \(5.9\).
In der Spalte Corr der Random effects steht die
Korrelation zwischen den zufälligen Schwankungen im Achsenabschnitt
und in der Steigung. Sie wird zu \(0.07\) geschätzt (95%-VI: \([-0.48; 0.68]\)). D.h., es gibt keine nennenswerte Korrelation zwischen der
Schwankung in dem Achsenabschnitt und der Schwankung in der Steigung
von Person zu Person (angenommen, die Korrelation wäre nahe bei 1
gewesen: Das würde bedeuten, dass grosse Werte von \(u_1\) zusammen mit
grossen Werten von \(u_2\) beobachtet werden; d.h., wenn jemand im
ausgeruhten Zustand eine überdurchschnittlich lange Reaktionszeit hat,
dann reagiert er besonders empfindlich auf Schlafentzug und die
Steigung ist grösser als im Populationsmittel).
Die \(u\)’s im Modell wurden beim fitten des Modells für jede Person
geschätzt (wenn man die Varianzen der zufälligen Effekte von Hand
ausrechnet, ist das Ergebnis leicht anders als die Varianzen im
summary-ouput, weil lmer eine andere Schätzmethode
verwendet):
## $Subject
## (Intercept) Days
## 308 2.2585509 9.1989758
## 309 -40.3987381 -8.6196806
## 310 -38.9604090 -5.4488565
## 330 23.6906196 -4.8143503
## 331 22.2603126 -3.0699116
## 332 9.0395679 -0.2721770
## 333 16.8405086 -0.2236361
## 334 -7.2326151 1.0745816
## 335 -0.3336684 -10.7521652
## 337 34.8904868 8.6282652
## 349 -25.2102286 1.1734322
## 350 -13.0700342 6.6142178
## 351 4.5778642 -3.0152621
## 352 20.8636782 3.5360011
## 369 3.2754656 0.8722149
## 370 -25.6129993 4.8224850
## 371 0.8070461 -0.9881562
## 372 12.3145921 1.2840221
##
## with conditional variances for "Subject"Für alle Personen gelten im Modell die fixen Effekte \(\beta_0 \approx 251.4\) ms und \(\beta_1 \approx 10.5\) ms/Tag. Bei Person
wurde eine individuelle Abweichung im Intercept von ca. \(2.3\) ms (etwas
langsamer als der Durchschnitt) und eine individuelle Abweichung in
der Steigung von ca. \(9.2\) ms/Tag (reagiert empfindlicher auf
Schlafentzug als der Durchschnitt) gefittet. D.h., für Person
308 sieht die modellierte Gerade so aus: \[y = (251.4 + 2.3) +
(10.5 + 9.2) \cdot \textrm{Days} = 253.7 + 19.7 \cdot \textrm{Days}.\]
Mit dem gefitteten Mixed Model können wir z.B. die Frage beantworten, wie unterschiedlich Personen auf Schlafentzug reagieren (mit der Standardabweichung der zufälligen Steigung, also ca. \(5.9\) ms; eine typische Schwankung von Person zu Person bzgl. Steigung ist also ungefähr \(6\) ms). Allerdings hat das Mixed Model auch Nachteile: Wir können z.B. nicht einfach ablesen, ob Person \(308\) im ausgeruhten Zustand eine signifikant grössere Reaktionszeit hat als z.B. Person \(335\). Für diese Frage wäre eine Lineare Regression mit einem Faktor für “Person” geeigneter. Zusammengefasst kann man sagen: Wenn man sich für die einzelnen Individuen der Studie interessiert, ist meist eine Lineare Regression mit Blockfaktoren günstig. Wenn man sich für die zu Grunde liegende Population interessiert, ist ein Mixed Model geeignet.
Die Vorhersage ist nun etwas komplizierter. Für eine konkrete neue
Person wissen wir z.B. nicht, wie gross ihre individuellen zufälligen
Effekte sind. Für schon beobachtete Personen haben wir zwar zufällige
Effekte geschätzt (wie oben bei Person 308 erklärt), aber es
ist nicht klar, wie genau diese zufälligen Effekte geschätzt wurden
und somit ist nicht klar, wie genau die Vorhersage ist. Vorhersagen
ohne Genauigkeitsangabe sind für beobachtete Personen allerdings
möglich (es werden dann die geschätzten zufälligen Effekte
verwendet). Welche Reaktionszeit sagt unser Modell z.B. für Person
308 nach 2.5 Tagen Schlafentzug voraus?
## 1
## 302.8293## identisch mit Berechnung "von Hand" (abgesehen von Rundungsfehler)
(251.405 + 2.257) + (10.467 + 9.199)*2.5## [1] 302.827Die Residuenanalyse funktioniert grundsätzlich wie bei der linearen Regression. Zusätzlich müssen wir nun noch prüfen, ob die zufälligen Effekte (in etwa) normalverteilt sind:
library(sfsmisc)
mult.fig(mfrow= c(2,2))
plot(fitted(fm), residuals(fm), main = "TA") ## Tukey-Anscombe Plot
qqnorm(residuals(fm)) ## QQ-Plot fuer Residuen
rEff <- ranef(fm) ## Zuf?llige Achsenabschnitt und Steigung pro Person
qqnorm(rEff$Subject[,1]) ## QQ-Plot von zuf. Achsenabschnitt
qqnorm(rEff$Subject[,2]) ## QQ-Plot von zuf. Steigung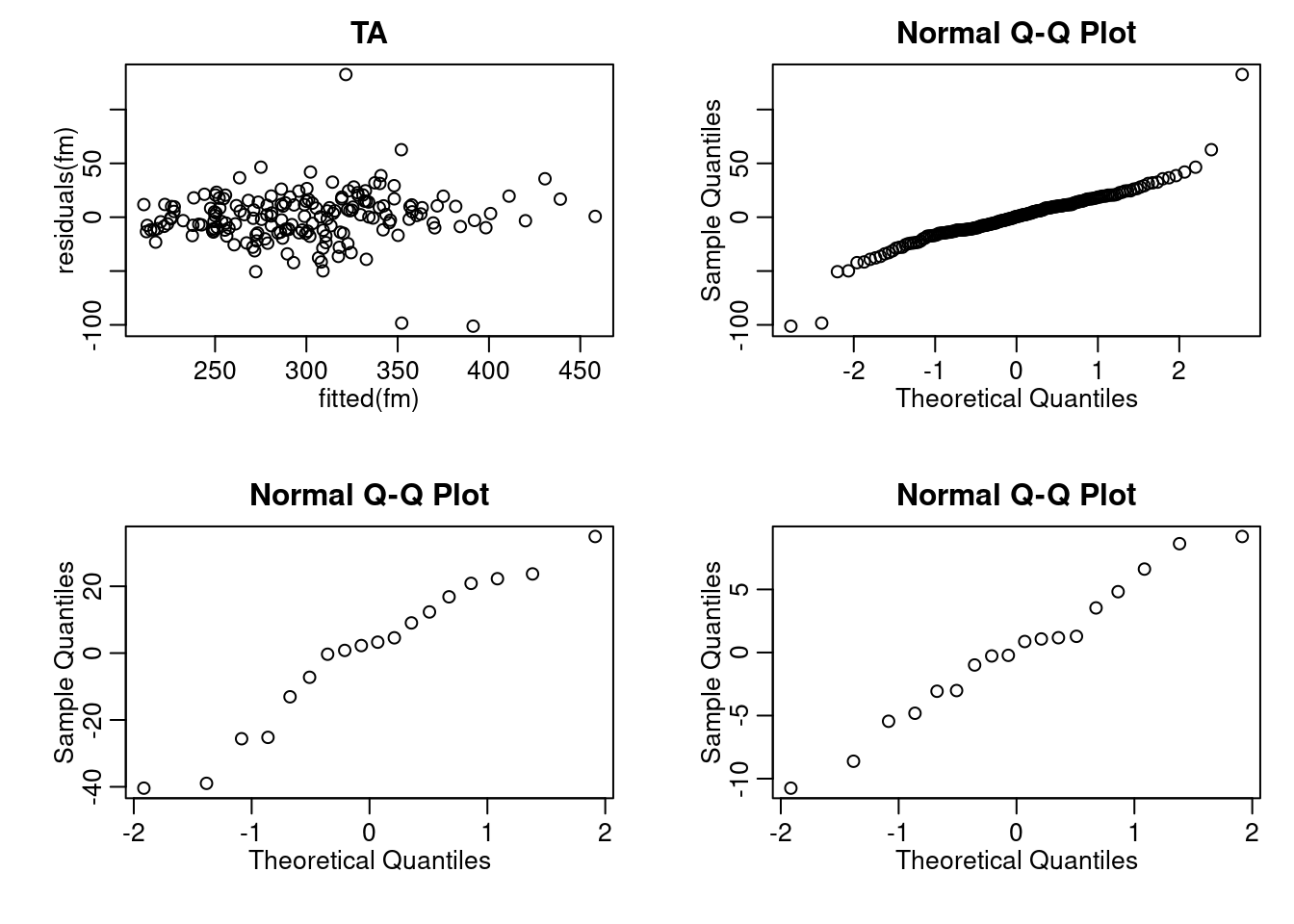
Umsetzung mit nlme
library(nlme)
fit <- lme(Reaction ~ Days, random = ~ 1 + Days | Subject, data = sleepstudy)
summary(fit)## Linear mixed-effects model fit by REML
## Data: sleepstudy
## AIC BIC logLik
## 1755.628 1774.719 -871.8141
##
## Random effects:
## Formula: ~1 + Days | Subject
## Structure: General positive-definite, Log-Cholesky parametrization
## StdDev Corr
## (Intercept) 24.740241 (Intr)
## Days 5.922103 0.066
## Residual 25.591843
##
## Fixed effects: Reaction ~ Days
## Value Std.Error DF t-value p-value
## (Intercept) 251.40510 6.824516 161 36.83853 0
## Days 10.46729 1.545783 161 6.77151 0
## Correlation:
## (Intr)
## Days -0.138
##
## Standardized Within-Group Residuals:
## Min Q1 Med Q3 Max
## -3.95355735 -0.46339976 0.02311783 0.46339621 5.17925089
##
## Number of Observations: 180
## Number of Groups: 18Wir erhalten die gleichen Resultate wie mit obigem Ansatz mit lme4.
Vertrauensintervalle erhalten wir hier mit der Funktion intervals.
## Approximate 95% confidence intervals
##
## Fixed effects:
## lower est. upper
## (Intercept) 237.927995 251.40510 264.88221
## Days 7.414662 10.46729 13.51991
## attr(,"label")
## [1] "Fixed effects:"
##
## Random Effects:
## Level: Subject
## lower est. upper
## sd((Intercept)) 15.5175354 24.74024072 39.4443767
## sd(Days) 3.9041696 5.92210282 8.9830375
## cor((Intercept),Days) -0.5760831 0.06556383 0.6572156
##
## Within-group standard error:
## lower est. upper
## 22.79706 25.59184 28.72925Diese Resultate unterscheiden sich leicht zu obigem Ansatz, weil verschiedene (statistische) Approximationen verwendet werden.